Ich habe eine Auswahl öffentlich verfügbarer Sequenzfragmente
(sog. "DNA-Barcodes") des mitochondrialen Gens Mt-CO1 (dieses
Gen codiert die Untereinheit I der Cytochrom-c-Oxidase) von
Tieren aus der Gattung Trichocera einer phylogenetischen
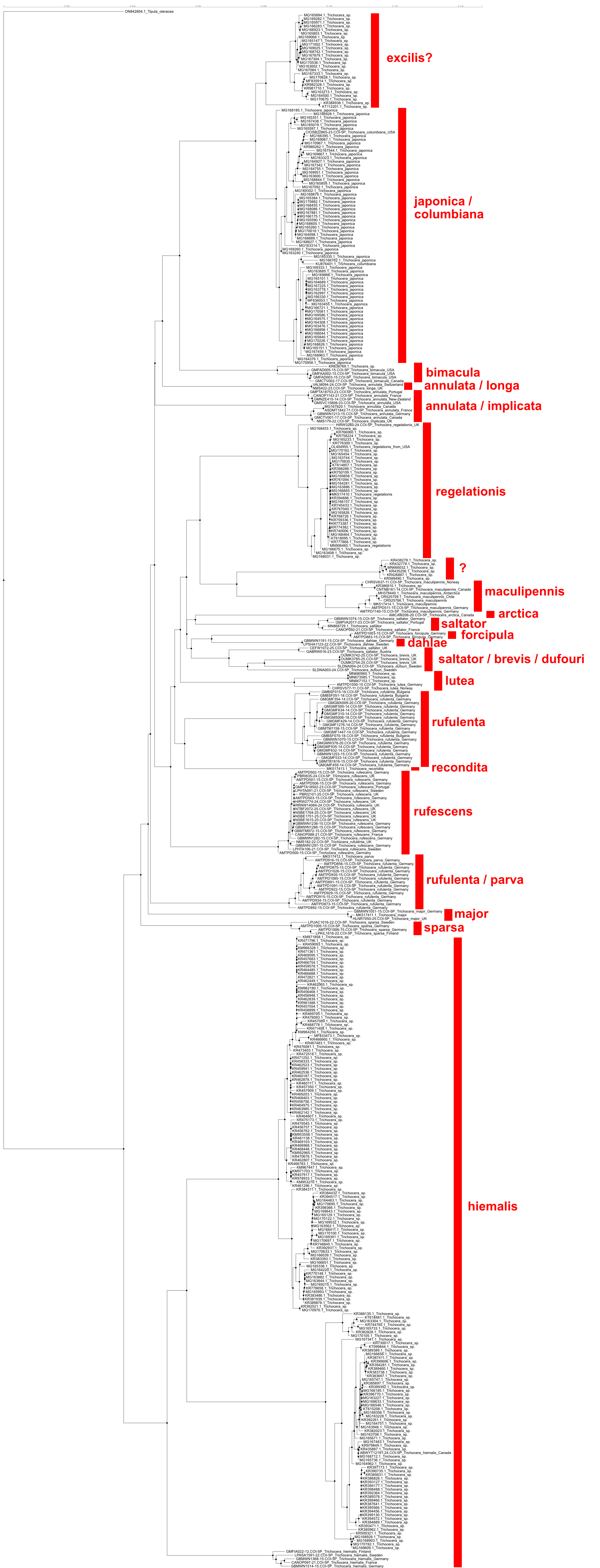
Analyse unterzogen (Abb. 1). Die Analyse zeigt zwei
grundsätzliche Aufzweigungen:
Gruppe 1: das Taxon hiemalis
Gruppe 2: alle übrigen Taxa
Diese Aufteilung und auch das weitere Verzweigungsmuster
innerhalb von Gruppe 2 entsprechen nicht der aktuell
verwendeten internen Strukturierung der Gattung in die
Untergattungen Trichocera, Metatrichocera, Saltrichocera, und
Staryia (siehe Krzeminska 2021). Ich gehe deshalb davon aus,
dass die Untergattungen keinen natürlichen Abstammungsgruppen
entsprechen und verwende sie nicht.
Gruppe 1 (= das Taxon hiemalis) zeigt intern eine erhebliche
Substrukturierung, vor allem in Nordamerika.
An der Basis der Gruppe 2 stehen die Taxa sparsa und major,
jeweils als monophyletische Gruppen. Darauf folgt ein
Monophylum aus Individuen, die als "parva" bzw. "rufulenta"
bestimmt wurden. Interessant ist, dass das Taxon rufulenta in
der Analyse zwei völlig separate Gruppen bildet. Ich vermute
deshalb, dass es bei der Bestimmung des Taxons Ungenauigkeiten
in der Identifikation gibt und deshalb oft Individuen des
Taxons parva als Taxon rufulenta bestimmt werden.
Möglicherweise sind die "rufulenta"-Individuen im Monophylum
"rufulenta/parva" fehlbestimmte Trichocera parva und die
zweite monophyletische "rufulenta"-Gruppe könnte dann das
eigentliche Taxon Trichocera rufulenta darstellen.
Es folgt in der Gruppe 2 eine erneute Aufteilung in zwei
größere monophyletische Gruppen:
Untergruppe A: aus den Taxa regelationis, maculipennis,
arctica, saltator, forcipula, dahlae, brevis, dufouri, lutea,
rufulenta, recondita und rufescens
Untergruppe B: aus den Taxa (vermutlich) excilis, japonica,
columbiana, bimacula, annulata, longa und implicata
In Untergruppe A bildet das Taxon rufescens (mit Ausnahme des
wahrscheinlich fehlbestimmten Individuums mit der
Zugriffsnummer NMS182-22.COI-5P (als Trichocera rufulenta
bestimmt)) eine monophyletische Gruppe.
Auch das Taxon recondita ist abgesetzt. Es bildet die
Schwestergruppe zu einem Monophylum aus als "rufulenta"
bestimmten Tieren. Wie oben erläutert, stellt diese Gruppe
möglicherweise das eigentliche Taxon rufulenta dar.
Es folgt das Taxon lutea als Monophylum. Kaum abgesetzt von
den übrigen Gruppen folgt dann ein Monophylum aus den Taxa
arctica, saltator, forcipula, dahlae, brevis und dufouri.
Intern zeigt sich diese Gruppierung jedoch problematisch: zwar
sind die Taxa arctica, forcipula und dahlae besser abgegrenzt
und erscheinen auch als Monophyla. Allerdings bildet das Taxon
saltator kein Monophylum und außerdem ist das Taxon dufouri
paraphyletisch in Bezug auf das Taxon brevis.
Interessanterweise spiegeln diese Probleme die bisherigen
taxonomischen Schwierigkeiten wider: Krzeminska (2021) weist
bereits darauf hin, dass das Taxon saltator aus zwei
morphologisch trennbaren Gruppen zu bestehen scheint: eine
Gruppe (die "echte" Trichocera saltator) deren Weibchen lange
Ovipositoren besitzen, und eine zweite Gruppe, deren Weibchen
kurze Ovipositoren besitzen und deshalb den Weibchen von
Trichocera brevis gleichen. Dies könnte die Ergebnisse der
phylogenetischen Analyse in Abb. 1 erklären, da hier ebenfalls
zwei "saltator"-Gruppen existieren: eine klar getrennte und
monophyletische "saltator"-Gruppe und eine weitere mit
"brevis" zusammen gruppierende "saltator"-Gruppe. In diese
zweite Gruppe fallen auch die beiden Sequenzen von Tieren, die
als "dufouri" bestimmt wurden. Diese bilden jedoch nur
zusammen mit den als "brevis" bestimmten Tieren ein
Monophylum. Die "dufouri"-Tiere könnten also fehlbestimmte
"brevis"-Tiere sein. Oder die Taxa brevis und dufouri sind
Synonyme. Als weitere Möglichkeit kommt jedoch auch in
Betracht, dass das Taxon brevis aus dem Taxon dufouri
hervorgegangen ist, denn in solchen Analysen auf
Individuenebene müssen Arten nicht monophyletisch erscheinen,
solange sie sich biologisch als zwei Arten verhalten (i. e.
sich nicht miteinander kreuzen). Hier besteht also noch
erheblicher Forschungsbedarf.
Schließlich bilden in Untergruppe A die Taxa regelationis und
maculipes klar abgegrenzte Monophyla. Als Schwestergruppe von
maculipes erscheint ein abgegrenztes Monophylum mit Individuen
aus Nordamerika, dem ich keinen Namen zuordnen kann, und das
ich deshalb in Abb. 1 mit einem Fragezeichen gekennzeichnet
habe.
In Untergruppe B bilden als "annulata" und "implicata"
bestimmte Tiere eine monophyletische Gruppe. Beide Taxa ähneln
sich sowohl im Bau der männlichen als auch der weiblichen
Genitalien sehr, so dass Fehlbestimmungen durchaus denkbar
sind. Auch an eine Synonymie der beiden Taxa kann gedacht
werden. Allerdings ist das Taxon annulata in der Analyse in
Abb. 1 nicht monophyletisch: ein "annulata"-Individuum aus der
Schweiz bildet eine weitere monophyletische Gruppe zusammen
mit einem als "longa" bestimmten Tier aus dem Vereinigten
Königreich von Großbritannien und Nordirland. Auch hier
besteht also noch Forschungsbedarf.
Ein weiteres abgegrenztes Monophylum bildet dann das Taxon
bimacula. Den Abschluss bildet ein Monophylum aus als
"japonica" bestimmten Individuen. Darin enthalten sind zwei
als "columbiana" bestimmte Individuen, möglicherweise
Fehlbestimmungen, da Krzeminska (2021) darauf hinweist, dass
Trichocera columbiana in der bisherigen Bestimmungsliteratur
schlecht charakteristiert ist und es deshalb häufig zu
Fehlbestimmungen kommt. Schließlich existiert noch ein
Monophylum aus nordamerikanischen Tieren als Schwestergruppe
zu den "japonica"-Individuen. Da dies also offensichtlich ein
"japonica"-verwandtes Taxon aus Nordamerika darstellt, könnte
es sich um das Taxon excilis handeln, das lange mit Trichocera
japonica synonymisiert wurde, nun aber als separates Taxon
aufgefasst wird (siehe Diskussion in Krzeminska 2021). Da dies
jedoch unsicher ist, habe ich das Taxon excilis in Abb. 1 mit
einem Fragezeichen versehen.
In dieser Analyse fällt sofort auf, dass die Beurteilung der
internen Strukturierung der beiden Hauptgruppen (Gruppe 1 und
Gruppe 2) sehr unterschiedlich ist. In Gruppe 1 wird trotz
offensichtlicher interner Strukturierung nur eine einzige Art,
nämlich Trichocera hiemalis, angenommen. In Gruppe 2 wird
einer ganz ähnlichen internen Strukturierung jedoch erheblich
mehr Aufmerksamkeit geschenkt und jede Linie als eigene Art
anerkannt. Es kann hinterfragt werden, ob eine solche
Ungleichbehandlung sinnvoll ist. Zwei Ansätze sind denkbar:
(a) die Linien in Gruppe 1 werden ebenfalls als eigene Arten
anerkannt und entsprechend beschrieben.
(b) der internen Substruktur in Gruppe 2 wird wie schon in
Gruppe 1 keine taxonomische Bedeutung beigemessen, und die
gesamte Gruppe 2 wird als eine Art mit dem ältesten
verfügbaren Namen aufgefasst: Trichocera regelationis
Linnaeus, 1758.
Es besteht hier also noch erheblicher Forschungsbedarf, der
neben weiteren genetischen Daten (z. B. Genomsequenzen) auch
die vielfach unbekannte Biologie der Taxa miteinbeziehen muss
(z. B. Genfluss-Analyses).
Madeira F, Madhusoodanan N, Lee J, Eusebi A, Niewielska A, Tivey ARN, Lopez R, Butcher S. 2024. The EMBL-EBI Job Dispatcher sequence analysis tools framework in 2024. Nucleic Acids Research 52(W1):W521-W525.
Ratnasingham S, Hebert PDN. 2007. BOLD: The Barcode of Life Data System (www.barcodinglife.org). Molecular Ecology Notes 7, 355-364.
Sayers EW, Bolton EE, Brister JR, Canese K, Chan J, Comeau DC, Connor R, Funk K, Kelly C, Kim S, Madej T, Marchler-Bauer A, Lanczycki C, Lathrop S, Lu Z, Thibaud-Nissen F, Murphy T, Phan L, Skripchenko Y, Tse T, Wang J, Williams R, Trawick BW, Pruitt KD, Sherry ST. 2022. Database resources of the national center for biotechnology information. Nucleic Acids Research 50(D1):D20-D26.
Sievers F, Higgins DG. 2021. The Clustal Omega Multiple Alignment Package. Methods in Molecular Biology 2231, 3-16.