Die Glyphipterigidae gehören innerhalb der Yponomeutoidea zu
einer Gruppe von Taxa, deren Rang (z. B. als Familia oder
Subfamilia) und Verwandtschaftbeziehungen untereinander unklar
sind. Üblicherweise werden die Plutellidae und die
Ypsolophidae als eigene Familien abgetrennt und die übrigen
Taxa im Rang von Unterfamilien (Orthoteliinae, Acrolepiinae,
Glyphipteriginae) als Familie Glyphipterigidae zusammengefasst
(Sohn et al. 2013). Ich folge dieser Auffassung hier in der
Zoographia Germaniae, allerdings deutet die phylogenetische
Analyse der inzwischen zahlreichen verfügbaren
Sequenzfragmente des Mt-CO1-Gens an, dass die Ypsolophidae
eine Gruppe innerhalb der Glyphipterigidae bilden und somit
als Unterfamilie in diese einbezogen werden müssten (Abb. 1).
Eine alternative Auffassung wäre es, alle bisherigen
Unterfamilien jeweils im Rang einer Familie anzuerkennen.
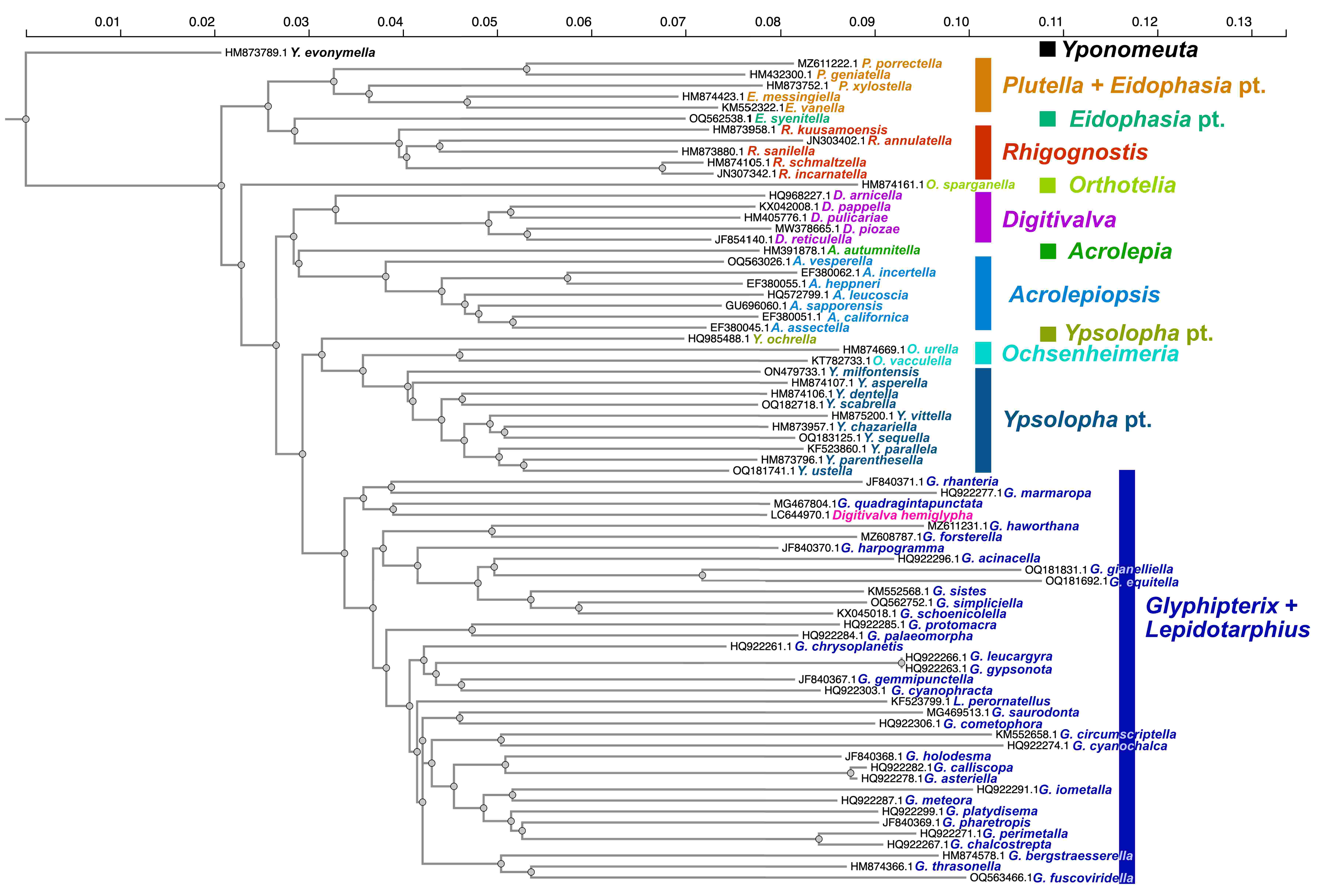
Abb. 1: Phylogenetischer Stammbaum
der Glyphipterigidae und verwandter Gruppen auf der
Grundlage der verfügbaren Sequenzfragmente des
mitochondrialen Gens Mt-CO1 (dieses Gen codiert die
Untereinheit I der Cytochrom-c-Oxidase). Der
Informationsgehalt des Sequenzfragments (658 bp, sog.
"DNA-Barcode") ist auf Grund der geringen Länge stark
begrenzt. Bei nahe verwandten Arten (wie in diesem Fall)
kann jedoch oft trotzdem ein phylogenetisches Signal
ausgewertet werden. Für den Stammbaum wurde die genetische
Distanz zwischen den Sequenzfragmenten errechnet (die Skala
zeigt den Umfang an genetischer Veränderung).
Distanzmethoden sind nur für DNA-Sequenzen aussagekräftig,
die nach der Molekularen Uhr evolvieren, was nur für wenige
Gene zutrifft, was aber für das Mt-CO1-Gen annähernd gegeben
ist. Grundlage der dargestellten Distanzbestimmung ist ein
Sequenzalignment mit Clustal Omega (Sievers & Higgins
2021; Madeira et al. 2024). Der Stammbaum wurde mittels der
Art Yponomeuta evonymella (Yponomeutidae) gewurzelt. Die
Buchstaben-Zahlen-Kombinationen, die im Stammbaum angegeben
sind, sind die Zugriffsnummern der jeweiligen Sequenzen in
GenBank (Sayers et al. 2022). Die Gattungen Plutella,
Eidophasia und Rhigognostis bilden eine monophyletische
Gruppe, die üblicherweise im Rang einer Familia
(Plutellidae) anerkannt wird. Auffällig ist, dass die
Gattung Eidophasia in der Analyse nicht monophyletisch ist,
was darauf hindeutet, dass die Gattungsgrenzen bislang
fehlerhaft definiert werden. Die Gattung Orthothelia zweigt
danach alleine ab; sie wird meist im Rang einer Unterfamilie
(Orthotheliinae) geführt. Die Gattungen Digitivalva,
Acrolepia und Acrolepiopsis bilden eine monophyletische
Gruppe, die üblicherweise im Rang einer Unterfamilie
Acrolepiinae (Halbmotten) der Glyphipterigidae geführt wird.
Manchen Autoren erkennen die Gruppe jedoch als eigene
Familie an, was nach der vorliegenden Analyse jedoch auch
zur Anerkennung einer Familie Orthoteliidae führen muss. Die
Gattungen Ypsolopha und Ochsenheimeria bilden eine
monophyletische Gruppe, die üblicherweise als Familie
Ypsolophidae geführt wird. In der vorliegenden Analyse ist
dies jedoch nur möglich, wenn auch die anderen Gruppen als
Familien anerkannt werden. Sollen Orthoteliinae und
Acrolepiinae weiterhin als Unterfamilien der
Glyphipterigidae geführt werden, so müssen die Ypsolophidae
als Unterfamilie in die Glyphipterigidae einbezogen werden.
Interessant ist die Stellung von Ypsolopha ochrella
außerhalb der übrigen Ypsolopha, was darauf hinweist, dass
diese nordamerikanische Art falsch zugeordnet ist und zu
einer anderen, möglicherweise neuen Gattung gehört. Die
Arten der Gattung Glyphipterix schließlich bilden eine klar
abgegrenzte monophyletische Gruppe, mit zwei Besonderheiten:
zum einen ist die Art Digitivalva hemiglypha (hervorgehoben
in Pink) offensichtlich in Digitivalva fehlplatziert und
gehört eigentlich in die Gattung Glyphipterix, und zum
anderen ist die Gattung Lepidotarphius offenbar ein Synonym
von Glyphipterix, da Lepidotarphius perornatellus inmitten
der Arten der Gattung Glyphipterix platziert wird.
Quellen und Einzelnachweise
Madeira F, Madhusoodanan N, Lee J, Eusebi A,
Niewielska A, Tivey ARN, Lopez R, Butcher S. 2024. The
EMBL-EBI Job Dispatcher sequence analysis tools framework in
2024. Nucleic Acids Research 52(W1):W521-W525.
Sayers EW, Bolton EE, Brister JR, Canese K, Chan J, Comeau DC,
Connor R, Funk K, Kelly C, Kim S, Madej T, Marchler-Bauer A,
Lanczycki C, Lathrop S, Lu Z, Thibaud-Nissen F, Murphy T, Phan
L, Skripchenko Y, Tse T, Wang J, Williams R, Trawick BW,
Pruitt KD, Sherry ST. 2022. Database resources of the national
center for biotechnology information. Nucleic Acids Research
50(D1):D20-D26.
Sievers F, Higgins DG. 2021. The Clustal Omega Multiple
Alignment Package. Methods in Molecular Biology 2231, 3-16.
Sohn JC, Regier JC, Mitter C, Davis D, Landry JF, Zwick A,
Cummings MP. 2013. A molecular phylogeny for Yponomeutoidea
(Insecta, Lepidoptera, Ditrysia) and its implications for
classification, biogeography and the evolution of host plant
use. PLoS One 8, e55066.
