
(1) Xylophagus ater Meigen, 1804
Archimyia atra (Meigen, 1804)
Erinna atra (Meigen, 1804)
Anmerkung: Die als "Xylophagus kowarzi" bestimmten Tiere bilden eine eigenständige Art. Allerdings scheint das Taxon "Erinna kowarzi" nomenklatorisch tatsächlich ein Synonym von Xylophagus ater zu sein (siehe weitere Informationen unter Xylophagus kowarzi).
Körperlänge etwa 10 bis 20 mm, wobei die Männchen deutlich
kleiner sind als die Weibchen. Die Art ist in Europa weit
verbreitet. Es werden keine Unterarten unterschieden.
Die Art ist an Laub- und Laubmischwälder oder Parks mit altem
Baumbestand gebunden. Die Imagines fliegen von April bis Mai.
Die Larven sind ausgewachsen etwa 2 bis 3 cm lang und
entwickeln sich tief in feuchtem, stark zersetztem Totholz von
Laubbäumen (Ulme, Buche, Linde, Birke, Zitterpappel). Sie
jagen dort andere totholzbewohnende Insektenlarven, stechen
sie mit den Mundwerkzeugen an und saugen sie vollständig aus.
Die Imagines nehmen vermutlich Pflanzensäfte an Saftflüssen
(z. B. an Stammwunden) auf.
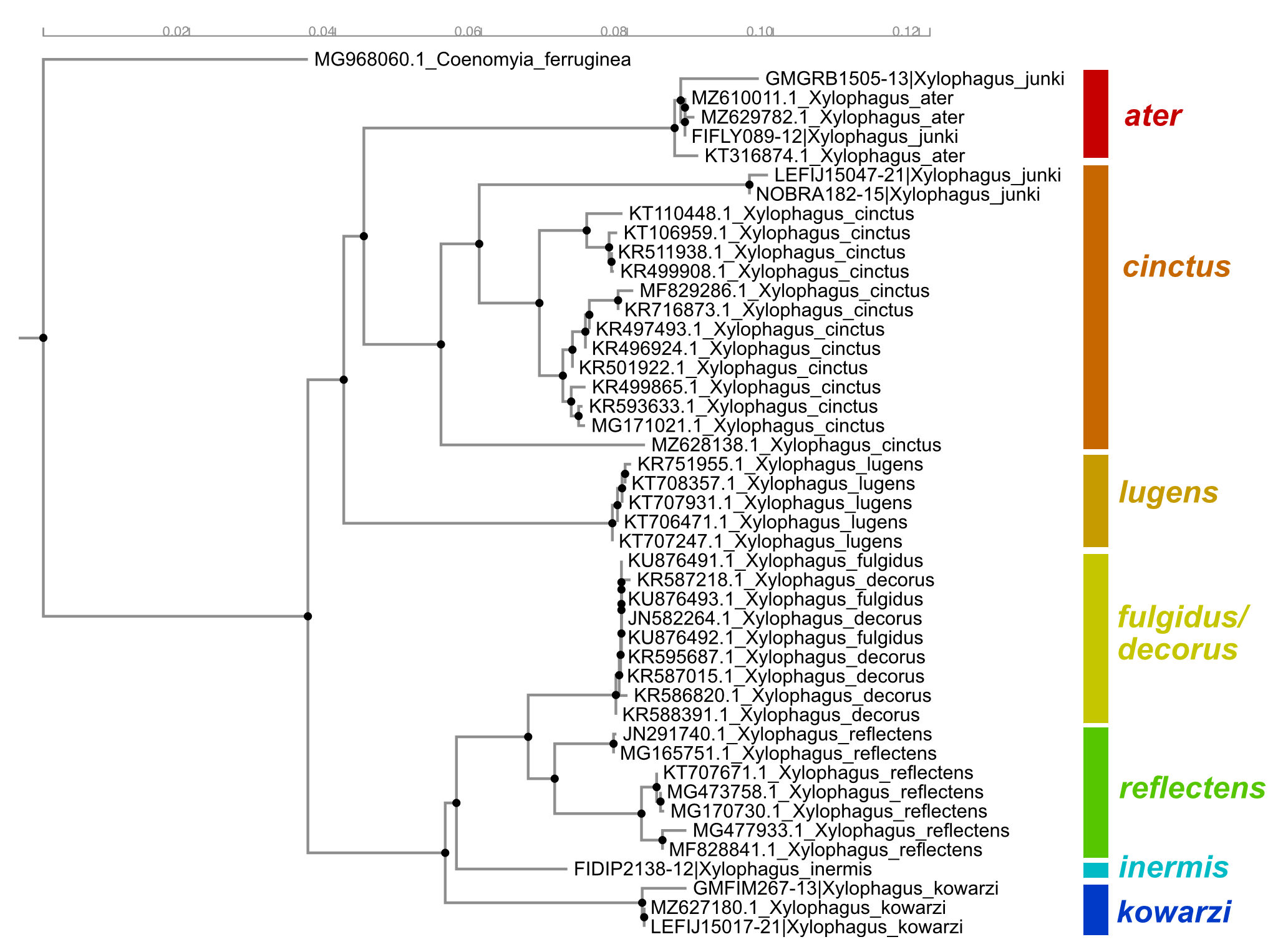
Nach der Analyse öffentlich verfügbarer Sequenzfragmente (sog.
"DNA-Barcodes") des mitochondrialen Gens Mt-CO1 (dieses Gen
codiert die Untereinheit I der Cytochrom-c-Oxidase), siehe
Abb. 1, sind die als Xylophagus kowarzi bestimmten Tiere nicht
konspezifisch mit Tieren, die als Xylophagus ater bestimmt
wurden, sondern bilden eine eigene monophyletische Gruppe
(weitere Informationen siehe Xylophagus
kowarzi). Stattdessen scheinen die als "Xylophagus
junki" identifizierten Individuen entweder falsch bestimmte
Xylophagus ater oder falsch bestimmte Xylophagus cinctus zu
sein.
Zur Nomenklatur: der Lectotypus von Xylophagus ater
wurde von Chandler, 1998 festgelegt, so dass das Taxon
nomenklatorisch eindeutig identifiziert ist. Der Status des
Taxons Xylophagus compeditus ist jedoch weiterhin ungeklärt
(siehe Xylophagus compeditus).
Krivosheina NP, Mamaev BM. 1966. Die Larven der europäischen Arten der Gattung Xylophagus Meigen (Diptera: Xylophagidae). Beiträge zur Entomologie 16, 275-283.
Krivosheina NP, Mamaev BM. 1988. Family Xylophagidae. In: Soos A, Papp L (Hrsg). Catalogue of the Palaearctic Diptera. Volume 5. Elsevier, Amsterdam, Oxford, New York, Tokyo. Pp 35-38.
Madeira F, Madhusoodanan N, Lee J, Eusebi A, Niewielska A, Tivey ARN, Lopez R, Butcher S. 2024. The EMBL-EBI Job Dispatcher sequence analysis tools framework in 2024. Nucleic Acids Research 52(W1):W521-W525.
Majer J. 1988. Family Coenomyiidae. In: Soos A, Papp L (Hrsg). Catalogue of the Palaearctic Diptera. Volume 5. Elsevier, Amsterdam, Oxford, New York, Tokyo. Pp 31-34.
Ratnasingham S, Hebert PDN. 2007. BOLD: The Barcode of Life Data System (www.barcodinglife.org). Molecular Ecology Notes 7, 355-364.
Sayers EW, Bolton EE, Brister JR, Canese K, Chan J, Comeau DC, Connor R, Funk K, Kelly C, Kim S, Madej T, Marchler-Bauer A, Lanczycki C, Lathrop S, Lu Z, Thibaud-Nissen F, Murphy T, Phan L, Skripchenko Y, Tse T, Wang J, Williams R, Trawick BW, Pruitt KD, Sherry ST. 2022. Database resources of the national center for biotechnology information. Nucleic Acids Research 50(D1):D20-D26.
Sievers F, Higgins DG. 2021. The Clustal Omega Multiple Alignment Package. Methods in Molecular Biology 2231, 3-16.
Woodley NE. 2011. A catalog of the World Xylophagidae (Insecta: Diptera). Myia 12, 455-500.
Zeegers T, Schulten A. 2022. Families of flies with three pulvilli. Field guide northwest Europe. Stichting Jeugdbondsuitgeverij, s´Graveland.